Customer success stories:
Rychlé sekvenování celého genomu (rWGS)
Specifikace projektu
Komplexní projekt rWGS sekvenování, realizovaný jako “proof-of-concept experiment”, byl navržen ve spolupráci s týmem prof. Milana Macka z 2. LF UK a FN Motol. Trio vzorků – rodiče a proband s diagnostikovaným Mayer-Rokitansky-Küster-Hauser syndromem (MRKH) – bylo vybráno skupinou prof. Macka s cílem ukázat potenciál a efektivitu celogenomového sekvenování (WGS) ve srovnání s analýzou sekvenačních panelů či exomu.
Celý proces byl navíc živě vysílán v průběhu konference DNA diagnostika 2023 konané ve dnech 20. – 21. března 2023 na Lipně.
Úvod
Rychlé celogenomové sekvenování, zkráceně rWGS (anglicky Rapid Whole Genome Sequencing) je průlomovou metodou zejména v případech, kdy klíčovým faktorem komplexní analýzy genomu je vedle přesnosti i rychlost. Ilustrativním případem použití může být testování novorozenců, u kterých je až 40 % úmrtí spojeno s extrémně rychlým průběhem zřídkavých genetických onemocnění.1-4 V klinické praxi může rWGS, ve srovnání s jinými diagnostickými přístupy (Tabulka 1), díky rychlé identifikaci kauzálních genetických variant pomoci zefektivnit léčbu nebo určit prognózu průběhu onemocnění.5
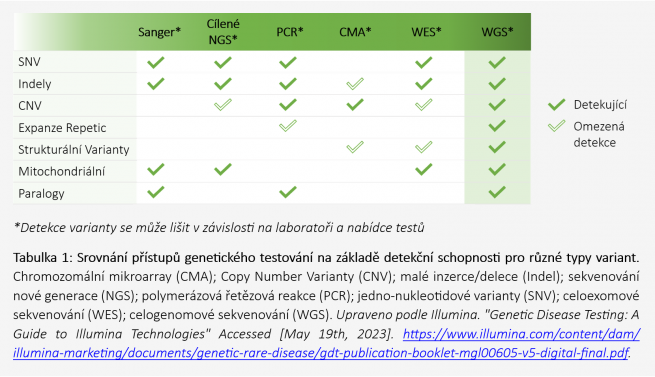
rWGS se zároveň stává zlatým standardem rychlé personalizované diagnostiky, neboť ve srovnání s jeho přínosy má velmi nízkou cenu, přičemž včasnou a přesnou identifikací kauzálních variant pomáhá mimo jiné snižovat i celkové náklady na zdravotní péči.6
Ve spolupráci s Institute of Applied Biotechnologies a.s. (IAB) představujeme rapid WGS protokol s výstupem vysoké kvality, který je postavený na NGS technologiích a nástrojích společností QIAGEN a Illumina. Optimalizovaný NGS protokol kombinuje nejpokročilejší produkty a přístupy izolace DNA, přípravy knihovny, sekvenování a datové analýzy následované interpretací anotovaných variant. Díky rWGS protokolu a genetické podpoře prof. Macka se podařilo zpracovat a vyhodnotit výsledky tria (probanda a jeho rodičů) za pouhých 39.9 hodin (Obrázek 1).
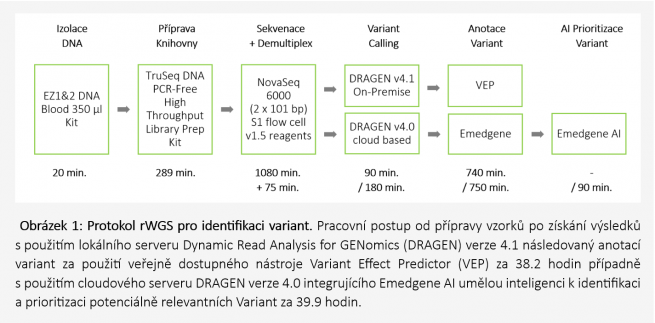
Metody
Purifikace DNA: Již během prvního kroku protokolu se podařilo dramaticky zkrátit čas přípravy použitím automatického izolátoru EZ2 Connect (QIAGEN), využívajícího principu purifikace magnetickými kuličkami. Genomická DNA (zkráceně gDNA) vysoké kvality byla izolována pomocí kitu EZ1&2 DNA Blood 350 µl Kit (QIAGEN) v průběhu 20 minut z malého objemu 3 vzorků krve tria (probanda a jeho rodičů).
Příprava knihovny: Celogenomové knihovny tria se vstupní koncentrací gDNA 1050 ng byly připraveny, naředěny a denaturovány, včetně provedení všech kroků kontroly kvality, během 289 minut za pomoci TruSeq DNA PCR-Free High Throughput Library Prep Kitu (Illumina). Použitý PCR-Free protokol představuje vynikající řešení pro špatně sekvenovatelné oblasti DNA, jakými jsou GC bohaté regiony, promotory nebo repetitivní úseky. Navíc použití mechanické fragmentace snižuje riziko tvorby nežádoucích artefaktů a mezer v knihovnách.

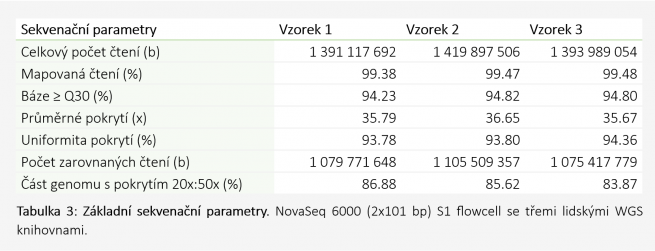
Sekvenace: rWGS sekvenování tria bylo provedeno na systému NovaSeq 6000 za použití S1 flowcelly (2 x 101 bp) (Illumina). Sekvenační experiment byl úspěšně dokončen za 18 hodin s datovým výstupem 464,92 Gb při 92.57 % čtení pokrytých více než 35x a 94.66 % čtení s Phred quality skóre vyšším než 30 (Q30). Všechny tři celogenomové knihovny hladce splnily požadovaný limit 800 miliónů párových čtení (paired-end) (Tabulka 3).
Analýza dat: Demultiplex sekvenačních dat s převodem CBCL do FASTQ byl proveden na cloudovém serveru BaseSpace Sequence Hub (Illumina) během 75 minut. Pro navazující kroky sekundární analýzy byly aplikovány dva různé přístupy.
První přístup využil pro zarovnání k referenčnímu genomu hg38 a volání variant lokální „Dynamic Read Analysis for GENomics“ (zkráceně DRAGEN) On-Premise server verze 4.1 (Illumina). Pro následnou anotaci identifikovaných variant byl použitý volně dostupný anotační nástroj „Variant Effect Predictor“ (zkráceně VEP) (Ensembl). Díky absenci limitů internetového připojení a plné kontrole výpočetních priorit trvalo vypracování reportu anotovaných variant ~14 hodin. Druhý přístup založený na cloudovém DRAGEN serveru verze 4.0 a softwarovém nástroji Emedgene (Illumina) dokončil anotaci variant za ~15.5 hodin. Nepatrně delší analýza druhého přístupu je však mnohonásobně zúročena skutečností, že namísto stovek netříděných anotovaných variant musí kliničtí specialisté projít jen několik klinicky relevantních variant předvybraných umělou inteligencí Emedgene (EmedgeneAI).
V našem případě Emedgene AI zařadila do užšího výběru 7 nejpravděpodobnějších kandidátních variant a dalších 63 možných variant, které byly zaslány k hodnocení laboratorním diagnostikům z 2. LF UK a FN Motol. Pro hlubší prozkoumání případu Emedgene disponuje funkcí rychlého filtrování, díky níž mohou klinici zobrazit exonové a intronové varianty v kandidátních genech, či se zaměřit na varianty s očekávanou segregací v rámci svých účtů u Emedgene.
Shrnutí
Tato stručná zpráva popisuje průlomový protokol rWGS, který integruje různé metodiky digitální genomiky včetně umělé inteligence do komplexního přístupu personalizované diagnostiky s cílem poskytnout prioritizované anotované varianty za méně než úctyhodných 40 hodin od odběru vzorku. Náš inovativní protokol založený na špičkových technologiích QIAGEN a Illumina, kombinující přesnost, rychlost a výjimečnou analytickou komplexnost, posouvá personalizovanou diagnostiku na úplně novou úroveň a otevírá možnosti použití lidské genetiky v klinické praxi. Prezentované výsledky ukazují, že rWGS je bezesporu jedním z nejpokročilejších řešení precizní diagnostiky s fantastickými výsledky v rychlé a přesné detekci kauzálních variant genetických poruch, kde ostatní diagnostické metody selhávají.
Velké poděkování a gratulace patří týmům profesora Milana Macka, IAB a našim kolegům z GeneTiCA Group, kteří se společně na celém projektu podíleli!
_______________________________________________________________________________________
1. Weiner J, Sharma J, Lantos J, Kilbride H. How infants die in the neonatal intensive care unit: trends from 1999 through 2008. Arch Pediatr Adolesc Med. 2011;165(7):630-634. doi:10.1001/archpediatrics.2011.102
2. Gunne E, McGarvey C, Hamilton K, Treacy E, Lambert DM, Lynch SA. A retrospective review of the contribution of rare diseases to paediatric mortality in Ireland. Orphanet J Rare Dis. 2020;15(1):311. doi:10.1186/s13023-020-01574-7Weiner
3. Berry MA, Shah PS, Brouillette RT, Hellmann J. Predictors of mortality and length of stay for neonates admitted to children's hospital neonatal intensive care units. J Perinatol. 2008;28(4):297-302.
4. Arth AC, Tinker SC, Simeone RM, Ailes EC, Cragan JD, Grosse SD. Inpatient Hospitalization Costs Associated with Birth Defects Among Persons of All Ages - United States, 2013. MMWR Morb Mortal Wkly Rep. 2017;66(2):41-46.
5. Petrikin JE, Cakici JA, Clark MM, et al. The NSIGHT1-randomized controlled trial: rapid whole-genome sequencing for accelerated etiologic diagnosis in critically ill infants. NPJ Genom Med. 2018;3:6. doi:10.1038/s41525-018-0045-8
6. Farnaes L, Hildreth A, Sweeney NM, et al. Rapid whole-genome sequencing decreases infant morbidity and cost of hospitalization. NPJ Genom Med. 2018;3:10. doi:10.1038/s41525-018-0049-4





1.png)




.png)

.png)

.png)

